Artigo 8: Automated identification of patient subgroups: A case-study on mortality of COVID-19 patients admitted to the ICU
A identificação de grupos sintomáticos é fundamental para abordagens macros no contexto médico, como por exemplos no tratamento de doenças endêmicas e pandêmicas. Neste sentido há um esforço do setor em utilizar a mineração de dados para esta tarefa afim de otimizar os tratamentos, auxiliar nas decições médicas de risco e possivelmente incrementar na curva de sobrevivência destes pacientes. Este artigo explora a aplicação de descoberta de subgrupos no contexto da COVID-19, procurando encontrar maneiras de automatizar a identificação dos pacientes que deram entrada na UTI em subgrupos.
O contexto da COVID-19
No âmbito das pesquisas clínicas, as análises de subgrupos envolvem a divisão de todos os pacientes em subgrupos, muitas vezes como meio de tornar populações heterogêneas mais homogêneas ou para responder a perguntas específicas sobre determinados grupos de pacientes, tipos de intervenção ou tipos de estudo.
No contexto da COVID-19, havia em contexto global uma situação crítica, com diversos hospitais superlotados com pacientes em diferentes níveis de riscos, com sintomas variados e históricos medicos diversos. A princípio, todos estes fatores se tornaram complicadores para a tarefa de encontrar padrões relevantes e de forma rápida, principalmente quanto os métodos tradicionais utilizados baseavam-se em análises manuais em limiares únicos. Além da ausência de informação concreta, em virtude da novidade da doença.
Nesse contexto, os métodos de Descoberta de Subgrupos (SD) foram relevantes para a análise de subgrupos clínicos, uma vez que estes métodos poderiam propor uma divisão automatizada dos dados em subgrupos complexos, ou seja, baseados em múltiplas variáveis e/ou múltiplos limiares.
Motivação e Objetivos
Então a partir da comoção e empenho global em torno da pandêmia os autores decidiram então buscar aplicar os métodos de Descoberta de subgrupos aos dados disponíveis com o objetivo de compreender se estes métodos automatizada=oa podem auxiliar na identificação de agrupamentos de pacientes de maneira relevante. Para isto, foram definidos dois críterios para avaliação dos resultados:
- Qualidades númericas: Verificando a coerência dos dados e evitando resultados advindos de acasos.
- Qualidades clínicas: Analisando a praticidade do uso destes dados para os especialistas clínicos.

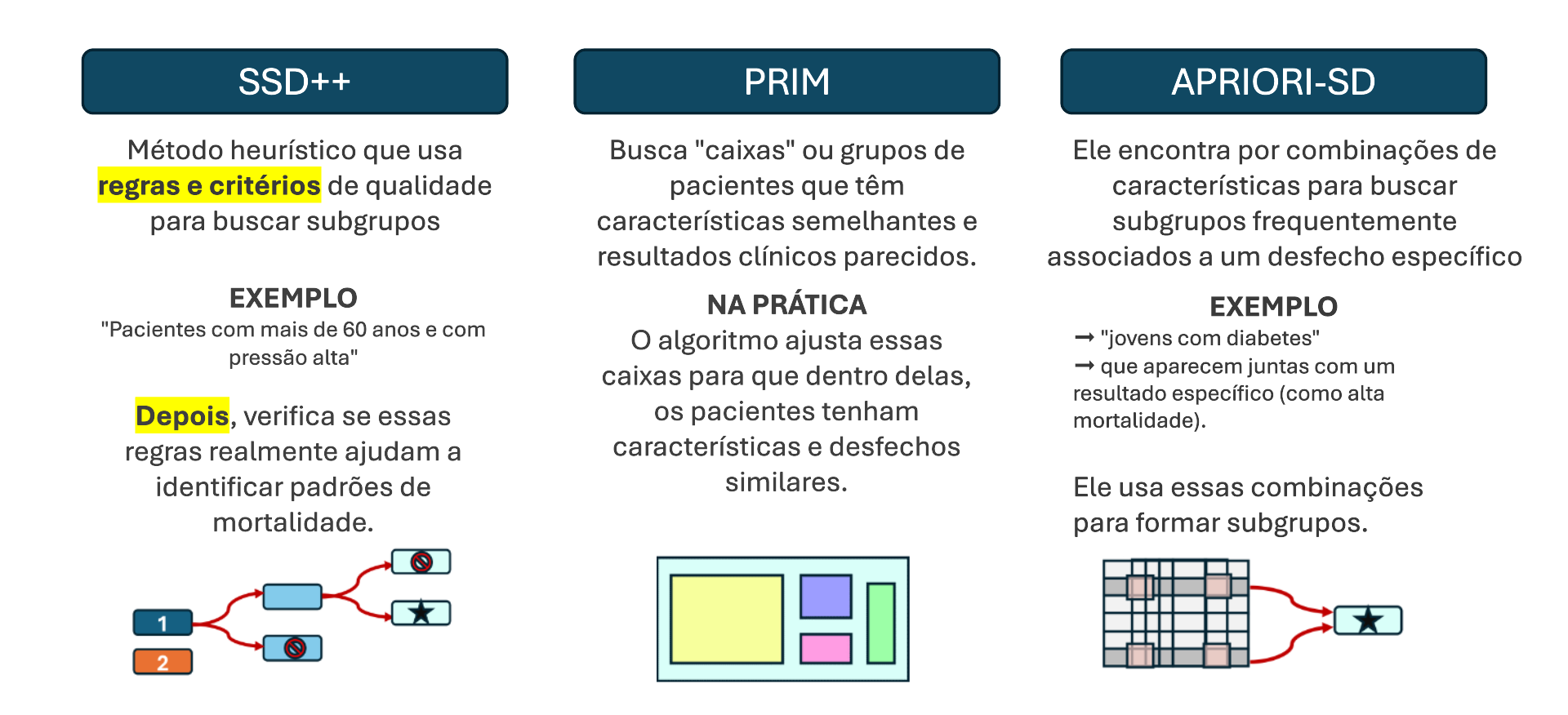
Além disso, três algoritmos de Descoberta de subgrupos foram estabelecidos como fonte de geração de resultados para as analises posteriores, sendo eles: SSD++, PRIM e APRIORI-SD.
Conjuto de Dados
Os dados utilizados para a análise são do Registro NICE (National Intensive Care Evaluation) na Holanda, abrangendo o período de fevereiro de 2020 a maio de 2022. O conjunto de dados inclui informações detalhadas sobre 14.548 pacientes internados em UTI, com 27,5% dos indivíduos falecendo durante a internação. As variáveis registradas incluem dados demográficos, valores laboratoriais e de monitoramento nas primeiras 24 horas de internação, diagnósticos, duração da internação e mortalidade hospitalar.
Durante o pré-processamento dos dados, foi realizada a manipulação de dados ausentes através da imputação múltipla por cadeias de equações (MICE), uma técnica que permite preencher valores faltantes. Além disso, foi feita a seleção de variáveis, resultando na exclusão de variáveis que apresentavam apenas um valor único, o que poderia comprometer a análise e a modelagem dos dados.
Aplicação
Os algoritmos de descoberta de subgrupos buscam encontrar regiões no espaço de busca com distribuições interessantes do atributo alvo. No trabalho em questão o atributo alvo é binário, representando o rótulo de diagnóstico para COVID-19. Para a realização desta tarefa, subgrupos foram considerados como combinações de restrições nos atributos das bases de dados, como por exemplo: (age >=25 and BMI < 19.)
Algoritmos
Dois tipos de algoritmos são explorados neste artigo, de busca exaustiva, envolve a exploração de todas as possíveis soluções ou caminhos para encontrar a solução ótima ou todas as soluções possíveis, e de busca heurística, que utiliza regras práticas ou intuições, para orientar a busca e encontrar soluções boas o suficiente sem explorar todas as possibilidades.
A principal vantagem de buscas exaustivas é a análise de todos os subgrupos possíveis, o que acaba gerando um alto custo computacional, para este método foi utilizado o algoritmo APRIORI-SD. Já a busca heurística possui um desempenho mais rápido e eficiente, mas como desvantagem tem sua precisão, exatidão e integridade reduzidas, além da baixa otimização, para esse método foi utilizado os algoritmos PRIM e SSD++.
- SSD++: Utiliza um método de busca heurística com Beam Search, onde a busca é guiada pela métrica de qualidade, a qual considera o critério Minimum Description Length (MLD). O algoritmo usa a estratégia Dividir e Conquistar, em cada iteração, e os dados cobertos por este subgrupo são removidos ou ponderados para orientar a busca. Também é realizada uma adição gulosa na busca, adicionando o subgrupo mais significativo em cada iteração.
- PRIM: Busca grupos que possuam características semelhantes e resultados clínicos parecidos, ajustando os algoritmos para que os pacientes de um mesmo grupo tenham características similares. Este algoritmo se assemelha a uma árvore de decisão. Elimina regiões menos propensas a retornar valores melhores para a métrica de qualidade (Peeling) e introduzindo pontos que favorecem valores melhores para a métrica de qualidade (Pasting).
- APRIORI-SD: Baseia-se em combinação de características para buscar subgrupos frequentemente associados em desfechos específicos, utilizando tais combinações para formar os subgrupos. Ele realiza a exporação do espaço de forma semelhante ao algoritmo de mineração de padrões frequentes Apriori, podando ramos com baixo suporte.
Metricas de qualidade e Scoring
Para além do funcionamento básico dos algoritmos supracitados, é necessário também o entendimento das métricas utilizadas pelos autores do artigo.
- Cobertura: Número de amostras corretamente classificadas sobre quantidade de amostras da classe alvo.
- Suporte: Número de amostras cobertas pelo subgrupo em relação ao total da base de dados.
- Comprimento de regra: Número de atributos seletores no subgrupo.
- Significância: Métrica estatística que determina se a diferença observada entre o subgrupo e a população é estatisticamente relevante.
- WRAcc: Avalia a utilidade de um subgrupo considerando tanto a cobertura quanto a precisão, ajustada para a distribuição de classe do conjunto de dados.
- Confiança: Número de amostras pertencentes a classe alvo sobre número de amostras não pertencentes a esta.
- Redundância: Seletores repetidos nos subgrupos retornados.
Classificação
Os autores fazem uso também de técnicas de classificação supervisionada, especificamente o modelo de regressão logística, para avaliar a qualidade dos subgrupos encontrados e se estes adicionam valor preditivo. Para poder realizar a classificação foram incluídos indicadores para cada subgrupos na base de dados original.
Métodos estatísticos foram utilizados para comparar o modelo base com o modelo estendido (teste ANOVA). Permitindo avaliar se a inclusão de uma variável indicadora de um subgrupo melhora, significativamente, a performance do modelo de classificação (usando relevância estatística de 0.05). Foi utilizada então a técnica de seleção stepwise (AIC) onde é adicionado e removido variáveis para encontrar o melhor ajuste do modelo.
Também foi realizada uma análise de significância clínica, comparando as descrições obtidas pelos subgrupos com os coeficientes de um modelo de regressão linear encaixado sob os dados de mortalidade no hospital. No entanto, esta segunda análise foi realizada usando uma seleção de variáveis backwards stepwise, que também usa o AIC.
Tudo isso foi realizado em conjunto a clínicos especialistas, que analisaram os subgrupos para validação dos resultados.
Metodologia
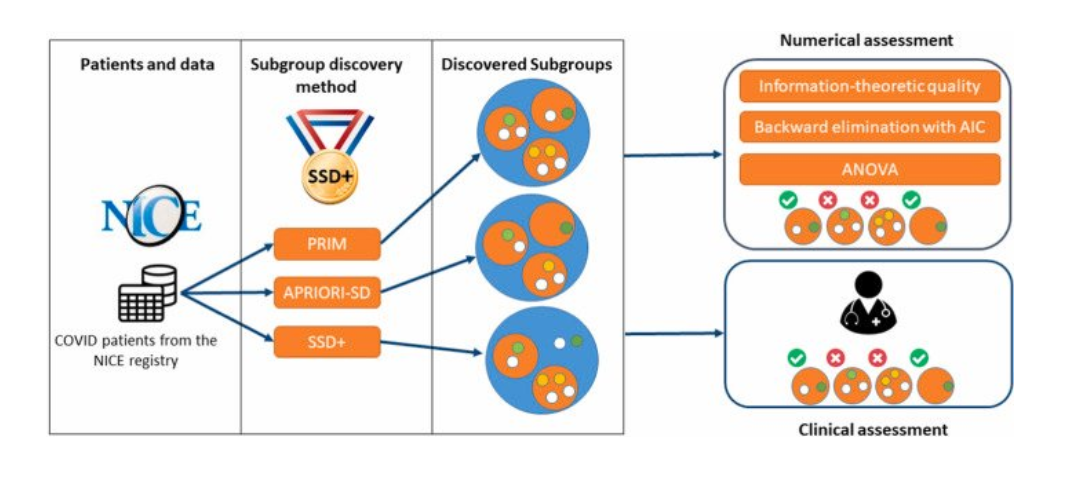
Como dito nos tópicos anteriores, os autores através das experimentações no contexto da COVID-19, desejam avaliar sistematicamente a qualidade numérica e clínica dos métodos utilizados: SSD++, PRIM e APRIORI-SD, com uma base dados em que há uma diversidade de pacientes com diferentes caractéristicas e níveis de risco.
A análise realizada envolveu a descoberta de subgrupos entre os pacientes com base em condições definidas pelos algoritmos supracitados. No qual, cada paciente foi alocado em um ou mias subgrupos conforme as regras e critérios estabalecidas pelos métodos. Os resultados obtidos por cada método foram analisados, tanto em termos de métricas quantitativas, como a precisão e a robustez dos subgrupos identificados, quanto em termos de relevância clínica, avaliando se os subgrupos encontrados possuíam significância prática para a gestão e o tratamento de pacientes.
Para garantir a eficácia dos modelos, os hiperparâmetros foram otimizados utilizando a técnica de grid search. Essa abordagem permitiu explorar diferentes combinações de parâmetros, como o número de subgrupos, a profundidade do algoritmo APRIORI-SD, e os parâmetros α e β do PRIM, além do número de candidatos para o SSD++. A otimização buscou maximizar o desempenho dos algoritmos em termos de qualidade dos subgrupos identificados.
A significância numérica dos subgrupos foi avaliada com base em métricas de qualidade e valor preditivo específicas para cada algoritmo, proporcionando uma medida objetiva da eficácia dos subgrupos na predição de resultados.
Adicionalmente, a significância clínica foi abordada de forma mais qualitativa. As descrições das regras dos subgrupos foram comparadas informalmente com os coeficientes de um modelo de regressão linear para verificar a consistência e a relevância clínica dos padrões identificados. Para complementar essa análise, dois intensivistas avaliaram a relevância clínica dos subgrupos encontrados, proporcionando uma perspectiva prática e especializada sobre a utilidade dos subgrupos na gestão e tratamento dos pacientes.
Resultados
Análise quantitativa dos resultados
Em relação aos aspectos quantitativos dos resultados obtidos pelos três algoritmos, pode-se notar que dentre as técnicas de descoberta de subgrupos o Apriori-SD foi o melhor em quase todas as métricas SD, com exceção da cobertura, que apresentou variação. Isso indica que este algoritmo teve um desempenho superior na identificação de subgrupos relevantes, exceto em termos de cobertura onde a variabilidade foi observada.
Relvância preditiva
Em termos de capacidade preditiva, a maioria dos subgrupos identificados sobreviveu ao processo de seleção stepwise, o que sugere que os subgrupos eram robustos e mantinham a sua relevância preditiva quando ajustados no modelo. Aproximadamente metade dos subgrupos identificados pelo Prim e pelo Apriori-SD continuaram a mostrar relevância preditiva significativa quando combinados com variáveis clínicas adicionais, mostrando assim a relevância dos algoritmos SD na identificação de padrões significativos e na melhoria da precisão preditiva dos modelos clínicos.
Analise qualitativa dos resultados
Na avaliação qualitativa, destaca-se no Apriori-SD todos os cinco subgrupos identificados foram considerados clinicamente significativos. Isso indica que o Apriori-SD foi altamente eficaz em encontrar subgrupos com relevância clínica clara e útil. O SSD++ demonstrou uma alta taxa de significância clínica, com 59,5 dos 62 subgrupos identificados sendo considerados clinicamente relevantes.
O SSD++ conseguiu identificar grupos com altas e baixas probabilidades de morte, mostrando a capacidade do método em discriminar entre diferentes níveis de risco, demonstrando grande diversidade nos subgrupos encontrados. Por outro lado, regras do tipo “não igual a”, comuns no PRIM, foram consideradas menos intuitivas e, portanto, menos úteis na prática clínica. Além disso, os grupos identificados pelo APRIORI-SD foram considerados menos distintivos em comparação com os subgrupos encontrados pelos outros métodos. Também foi notado que algumas variáveis incluídas na análise se mostraram clinicamente não significativas, o que pode ter impactado a eficácia dos modelos na prática clínica.
Aplicações semelhantes
Existem aplicações similares à abordagem feita no artigo, como por exemplo o Sonoconsult, um sistema de ultrassonografia abdominal integrado com técnicas de mineração de dados e estatística. A abordagem utiliza o software Vikamine em um hospital alemão desde 2005. Os principais benefícios do seu uso são a construção de relatórios completos e padronizados, auxiliando na documentação dos resultados, alem de alta acurácia no diagnóstico, facilitando também o trabalho de médicos iniciantes. As referências estão disponíveis nos artigos: Exploiting Background Knowledge for Knowledge-Intensive Subgroup Discovery, Semi-Automatic Visual Subgroup Mining using VIKAMINE e Application and Evaluation of a Medical Knowledge-System in Sonography(SonoConsult).
Dessa forma, a abordagem de analisar subgrupos de pacientes também se expande para outras áreas da medicina. Como exemplo, para aplicações em doenças cardíacas, identificando grupos de paciente com maior risco para problemas do coração ( Expert-Guided Subgroup Discovery: Methodology and Application, Active Subgroup Mining: A Case Study in Coronary Heart Disease Risk Group Detection, Subgroup Discovery for Actionable Knowledge Generation: Shortcomings of Classification Rule Learning and the Lessons Learned), além da verificação da qualidade de registros médicos, visto que dado a quantidade de pacientes, os registros podem acabar sendo feitos de maneira inadequada, sendo necessário analisar o que indica boas documentações clínicas, como disponível no artigo Profiling Examiners using Intelligent Subgroup Mining.
Discussões
Atualmente, devido a recente pandêmia de COVID-19 diversos estudos na aréa da saúde, como este relatado no artigo analisado. Para se ter uma ideia, mais de 700 estudos de predição diagnósitca e prognóstica de COVID-19 foram produzios neste período, retornando modelos para diversas condições de saúde com diferentes resultados. Tendo em vista essa grande quantidade de informação, uma preocupação é a qualidade desses modelos, sendo necessário estudos e verificações para garantir e reproduzir a relevância desses métodos, além do uso cuidadoso dos modelos disponibilizados.
Além disso, outos tópicos a serem debatidos são referentes a segurança, privacidade e tratamento dos dados coletados. Como muitas dessas aplicações médicas necessitam de informações dos pacientes, dados de saúde e condições clínicas, surgem mecanismos de evitar com que esses dados sejam divulgados ou utilizados de maneira inadequada, como é o caso da Equator. A Equator é uma iniciativa internacional que busca melhorar a confiabilidade dos dados presentes em publicações e literatura médica, promovendo a verificação e transparência dos trabalhos, definindo guias de utilização dessas informações.
Replicando a obtenção de resultados
Base de dados
A base de dados disponibilizada no artigo está presente no link a seguir: https://www.stichting-nice.nl/extractieverzoek.jsp. A base de dados tem sua execução baseada no formato ‘feather’, sendo um formato binário utilizado para armazenar informações de maneira rápida e eficiente, útil para o trabalho com grande quantidade de informações, além de ser suportado por diferentes linguagens de programação.
Apesar do link ser disponibilizado no artigo, ela não é publicamente acessível, sendo necessário realizar uma solicitação de acesso, o que pode demorar algumas semanas. Dessa forma, a reprodução e testes com os dados utilizados no trabalho não foi possível de ser realizada. Contudo, ainda é possível comentar sobre a implementação e execução dos testes conforme mencionado no artigo.
Repositório dos Algoritmos
Em relação aos algoritmos mencionados, é possivel encontra-los facilmente em repositórios e bibliotecas públicas. Segue a então a lista de referências para estes: - Apriori-SD: Sua implementação é baseada na biblioteca Python pysubgroup, com a documentação disponível em https://pysubgroup.readthedocs.io/en/latest/ . - PRIM: Tem sua implementação e documentação disponível em https://github.com/martinsps/PRIM. - SSD++: Possui sua implementação baseada no SSDpp-numérico, disponibilizado no repositório: https://github.com/HMProenca/SSDpp-numeric.
Execução
No procedimento de execução, há duas rotas possíveis de se explorar. A primeira rota é a execução dos algoritmos em cima do dataset, e a segunda é o procedimento de comparação entre os três metodos. Ambas as rotas são feitas através do script python subgroups.py referenciado no artigo. Para executá-lo, é apenas utilizar este formato de comando no terminal:
python subgroups.py [dataset] [modo]Neste caso, o [dataset] deve ser trocado pelo arquivo contento os dados a ser utilizados na analise. Já o [modo] pode possuir apenas dois valores: algorithm e compare. No modo de algorithm, um terceiro argumento é adicionado a linha de comando sendo este, o nome do algoritmo a ser utilizado (prim, apriori, beam e ssdpp). Um exemplo de comando seria:
python subgroups.py data.feather algorithm aprioriJá no modo compare, nenhum novo argumento é adicionado ficando apenas:
python subgroups.py data.feather compareConclusão
Concluindo, o artigo foca então não em fazer uma análise médica referente a COVID com os resultados obtidos, mas sim mostrar uma metodologia de análise e tratamento de dados e como isso pode ser aplicada em outros ramos da saúde. O trabalho desenvolvido é executado de forma a encontrar os subgrupos mais relevantes, que são removidos continuamente para validar o modelo construído, construindo assim uma lista de subgrupos que geram os melhores modelos de regressão para o risco envolvido na doença.
Assim, os algoritmos testados possuem diferentes compromissos, com o Apriori retornando bons resultados nas métricas de qualidade, no entanto, atrelado a subgrupos genéricos e redundantes. Já o SSD por exemplo possui o foco de encontrar mais subgrupos e utilizar descrições mais diversas, focando então em diminuir a redundância, porém sem utilizar grupos tão complexos, também retornando boas métricas de qualidade e resultados com validade clínica para serem analisados pelos médicos.
Referências
VAGLIANO, I.; KINGMA, M.Y.; DONGELMANS, D.A; de LANGE, D.W.; de KEIZER, N.F.; SCHUT, M.C. Automated identification of patient subgroups: A case-study on mortality of COVID-19 patients admitted to the ICU. Disponível em: https://www.sciencedirect.com/science/article/pii/S001048252300611X?via%3Dihub
ATZMUELLER, M. Semi-Automatic Visual Subgroup Mining using VIKAMINE. Disponível em: https://pdfs.semanticscholar.org/b163/6e7ae20a6bf7db347b942a5480b00ac2bb70.pdf.
ATZMUELLER, M.; PUPPE, F.; BUSCHER, H.-P. Exploiting background knowledge for knowledge-intensive subgroup discovery. Disponível em: https://www.ijcai.org/Proceedings/05/Papers/1217.pdf. Acesso em: 27 jul. 2024a.
ATZMUELLER, M.; PUPPE, F.; BUSCHER, H.-P. Profiling Examiners using Intelligent Subgroup Mining. Disponível em: https://file.biolab.si/biolab/idamap/idamap2005/papers/17%20Atzmueller%20CR.pdf.
GAMBERGER, D.; LAVRAC, N. Expert-guided subgroup discovery: Methodology and application. 2002. Disponível em: http://arxiv.org/abs/1106.4576.
GAMBERGER, D.; LAVRAČ, N.; KRSTAČIĆ, G. Active subgroup mining: a case study in coronary heart disease risk group detection. Artificial intelligence in medicine, v. 28, n. 1, p. 27–57, 2003.
PUPPE, F. et al. Application and evaluation of a medical knowledge system in sonography (SONOCONSULT). Proceedings of the 2008 conference on ECAI 2008: 18th European Conference on Artificial Intelligence. Anais…NLD: IOS Press, 2008.