Artigo 1: Differentiable Pattern Set Mining
1 Introdução: o que é mineração de padrões?
Mineração de padrões é uma área de Aprendizado Descritivo que objetiva encontrar informações interpretáveis de grandes bancos de dados. Tal objetivo é alcançado por meio da mineração de “padrões” ou “regras” que definem conjuntos relevantes, seja em frequência ou em importância.
As aplicações de mineração de padrões são variadas, desde ciências naturais como Biologia e Química, estudos estatísticos e matemáticos e análises societais e comportamentais.
Infelizmente, os algoritmos para mineração de padrões são extremamente custosos, sendo duplamente exponencial no número de atributos. Além disso, eles sofrem de redundância nas respostas, muitas vezes repetindo padrões similares e de relevância das respostas, sofrendo com overfitting ou underfitting.
2 BinaPs
Para resolver a problemática acima, o algoritmo introduz o BinaPs, uma solução que vê o problema por meio da lente de aprendizado de máquina ao invés de construção de conjuntos. Para entender melhor as duas perspectivas, considere as diferenças principais do BinaPs para algoritmos tradicionais:
- Apriori, Eclat, FP-Growth:
- Padrões escolhidos por frequência
- Padrões redundantes encontrados
- Apenas otimizados por heurísticas
- Calculado na CPU
- BinaPs:
- Funções diferenciáveis para descoberta de padrões
- Padrões menos redundantes
- GPU para cálculos
- Significativamente mais escalável
- Otimizável por parâmetros de entrada como learning rate
2.1 Funcionamento
Inspirado por decoders e encoders, o algoritmo tem como objetivo obter os atributos de um item, codificar eles em o mesmo número ou menos de neurônios no processo e retornar para o número inicial como representado pela Figura 1.
O BinaPs contém algumas otimizações e considerações quando considerado com outras rede neurais, dentre elas:
- Os neurônios e pesos finais são binários \(\{0, 1\}\)
- Viés é usado para evitar overfitting
- Normalização da função de perda para evitar saídas de apenas \(\{1\}\) ou \(\{0\}\) em bases densas e esparsas.
- Gated Straight-Through Estimator para não penalizar os neurônios desligados
Após essas mudanças, o algoritmo funciona como descrito pela Figura 2.
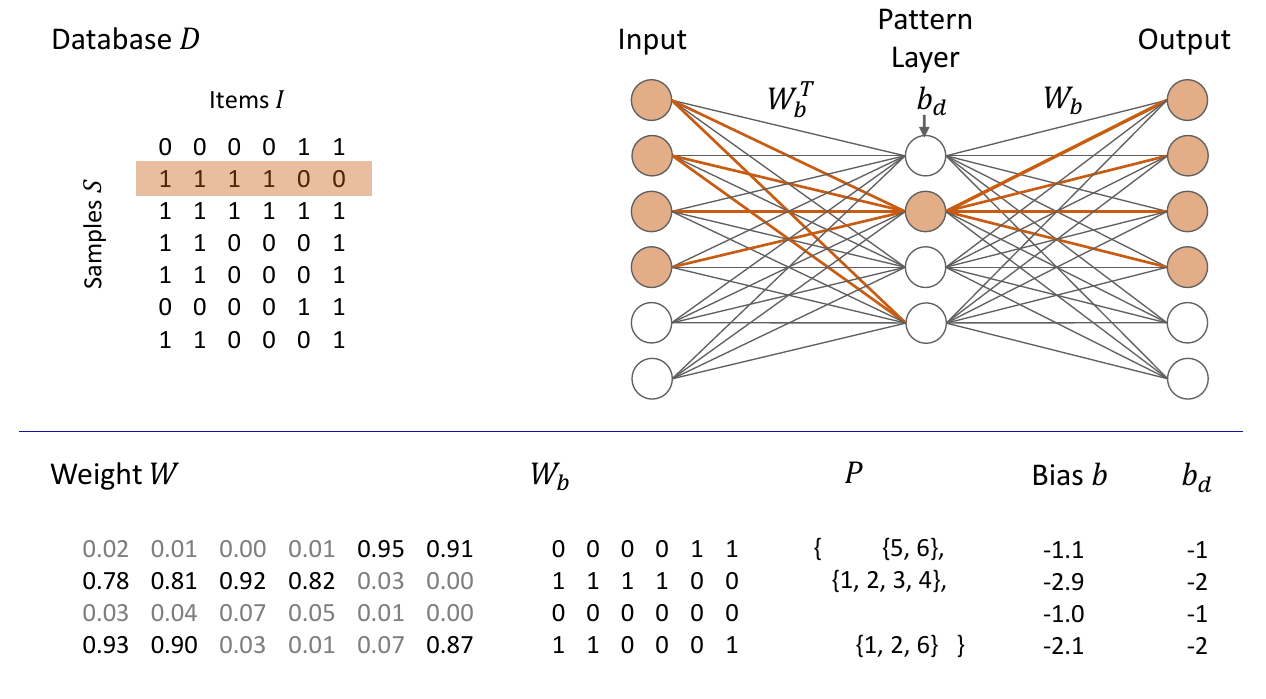
2.2 Experimentos
O BinaPs foi comparado com três outros competidores na área de mineração de dados: Asso, Slim e Desc, que usam matrizes booleanas, mineração de conjuntos por MDL e maximum entropy modeling respectivamente. Os experimentos foram divididos em duas categorias: sintéticos e reais.
2.2.1 Sintéticos
Dados sintéticos foram escolhidos pois é possível inserir ou estudar padrões reais nos dados previamente, podendo assim comparar os resultados dos algoritmos com o “ground truth”, ou seja, o que assumimos ser a verdade.
Para medir a performance dos algoritmos foi usado a F1-score, uma medida calculada pela média harmônica do precision e do recall, comumente usada em aprendizado de máquina para avaliar a performance de modelos.
2.2.1.1 Escalabilidade
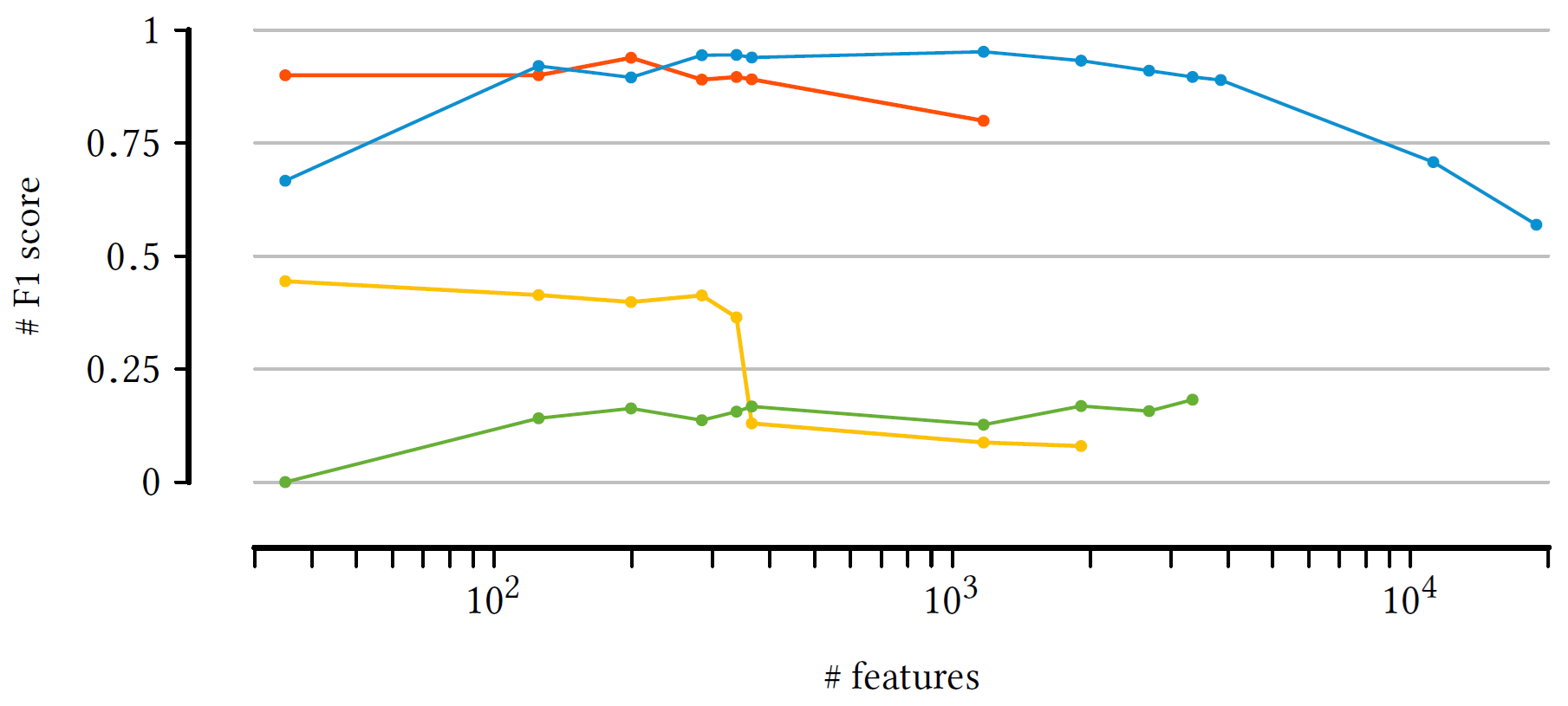
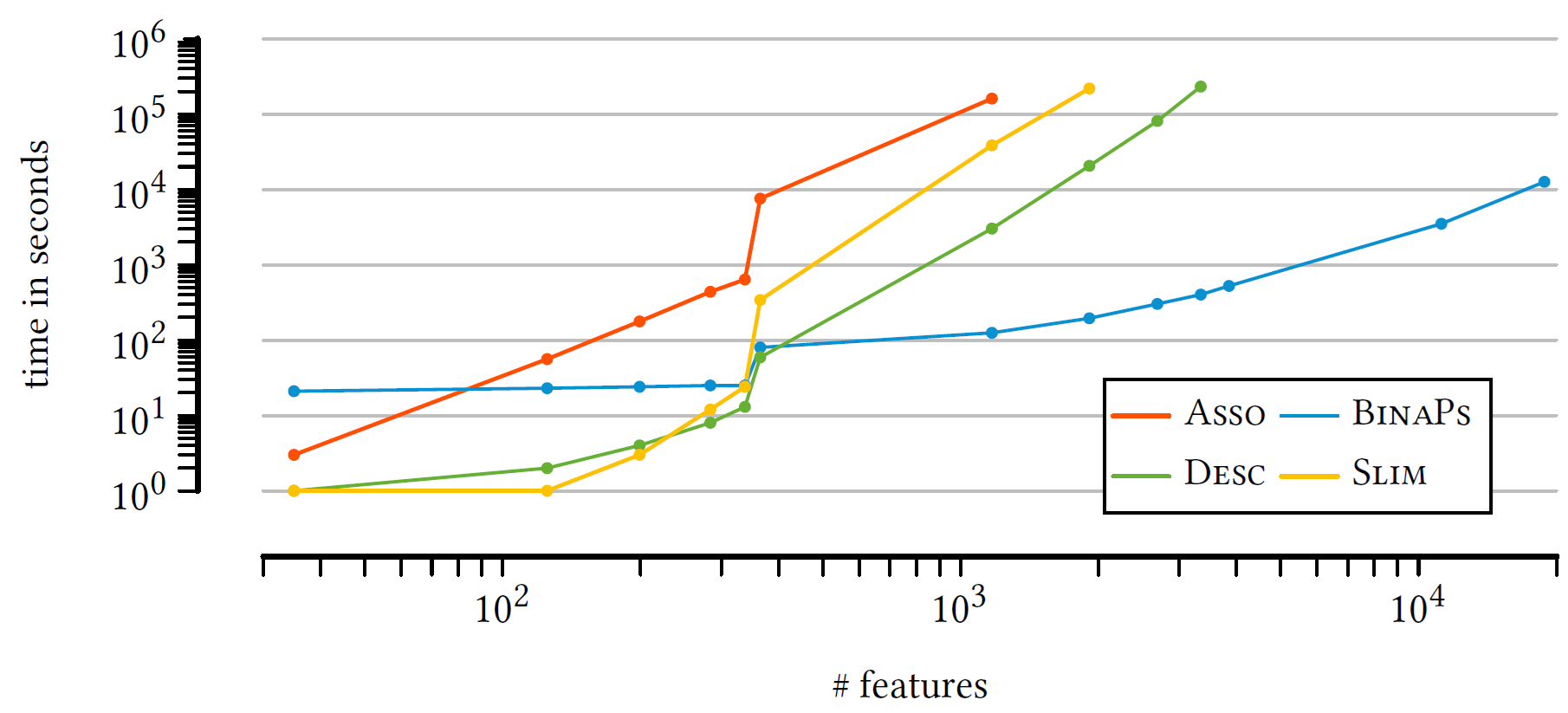
Com um número crescente de features (atributos), o BinaPs se mostra mais preciso (Figura 3 (a)) e menos custoso (Figura 3 (b)) em tempo que os outros três, com o Asso se assemelhando a ele em resultados, mas não em performance, eventualmente não sendo capaz de rodar datasets maiores.
2.2.1.2 Resistência a ruídos
Muitas vezes dados reais possuem ruídos, seja de medidas errôneas, problemas no dataset ou imprevisibilidade dos dados. Para testar se os algoritmos são resistentes a tais cenários foram inseridos quantidades crescentes de ruídos nos databases testados com os seguintes resultados:
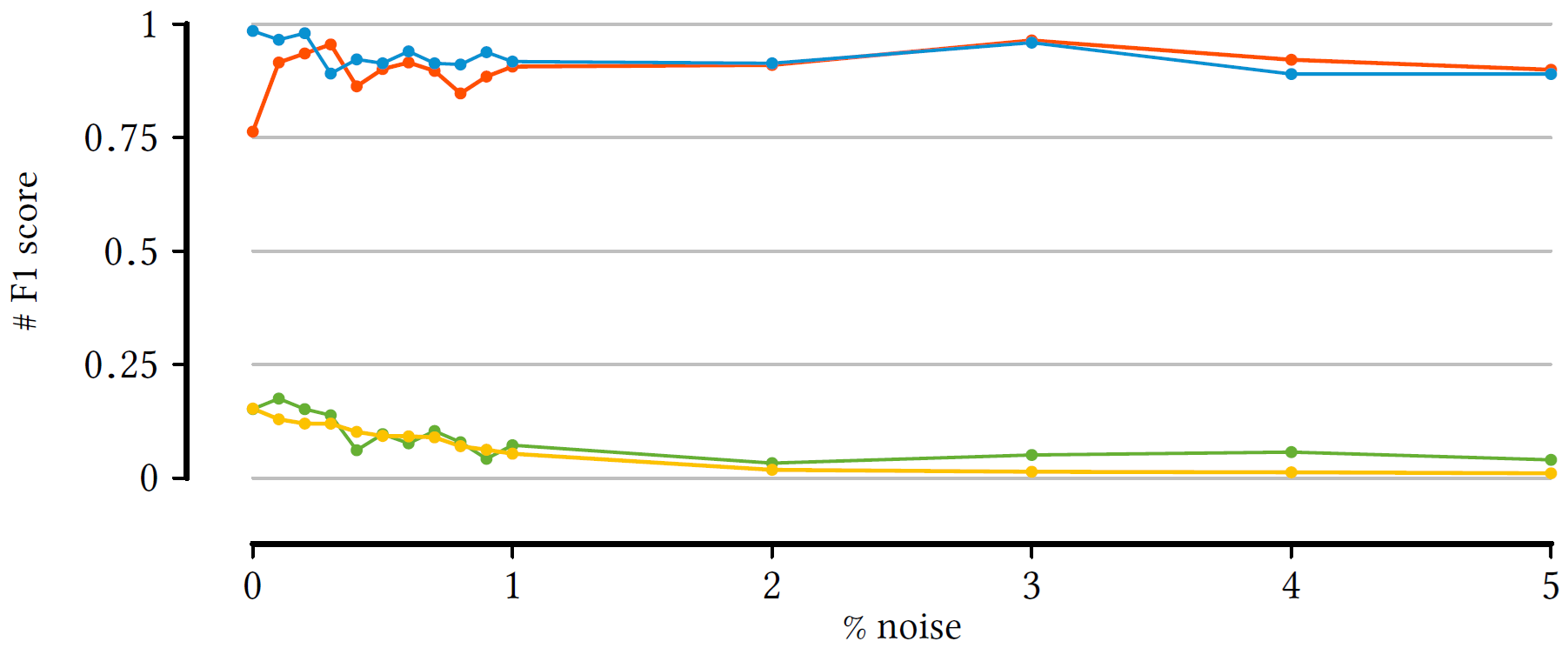
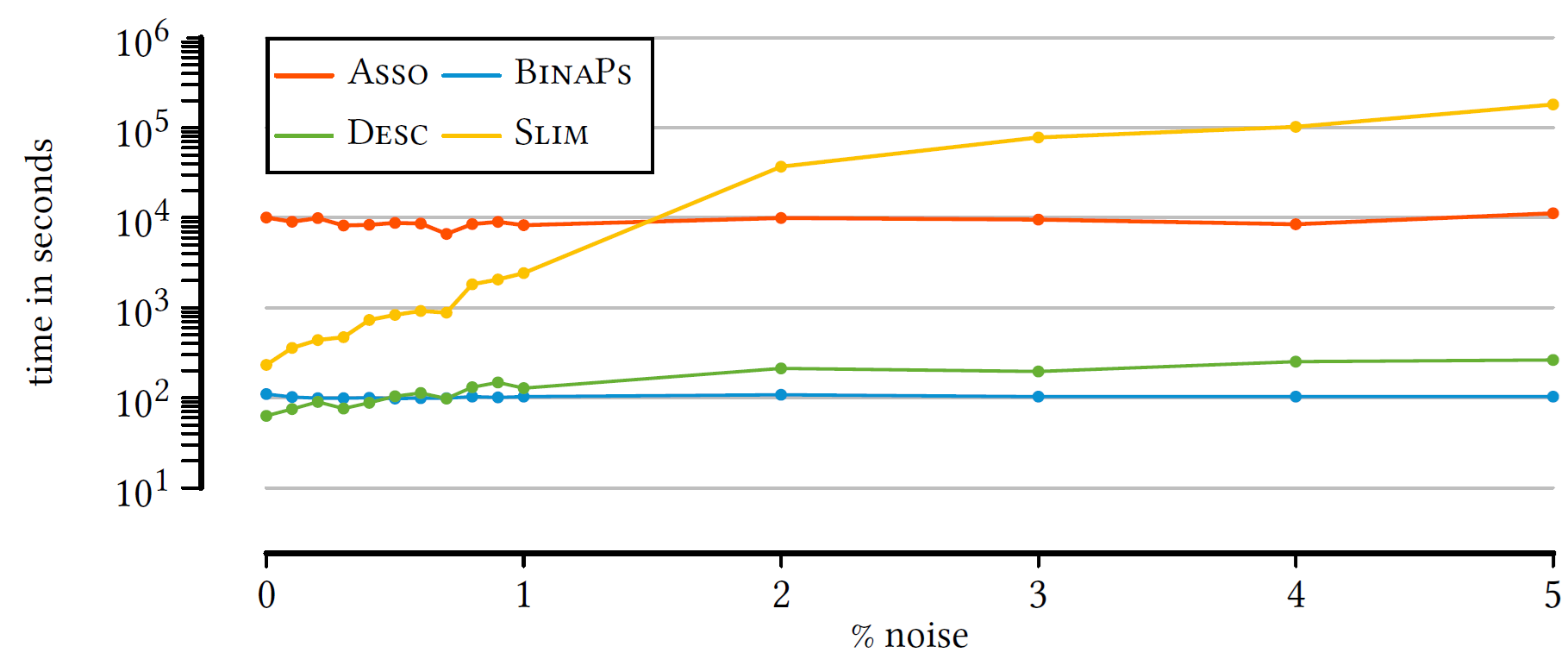
Ao analisar os gráficos, ambos o BinaPs e o Asso são resistentes a ruídos em ambos F1-score (Figura 4 (a)) e tempo de execução (Figura 4 (b)). Já ambos Slim e Desc são afetados por ruídos no F1-score (Figura 4 (a)) e o Slim em tempo de execução (Figura 4 (b)) também.
2.2.1.3 Operabilidade com Samples
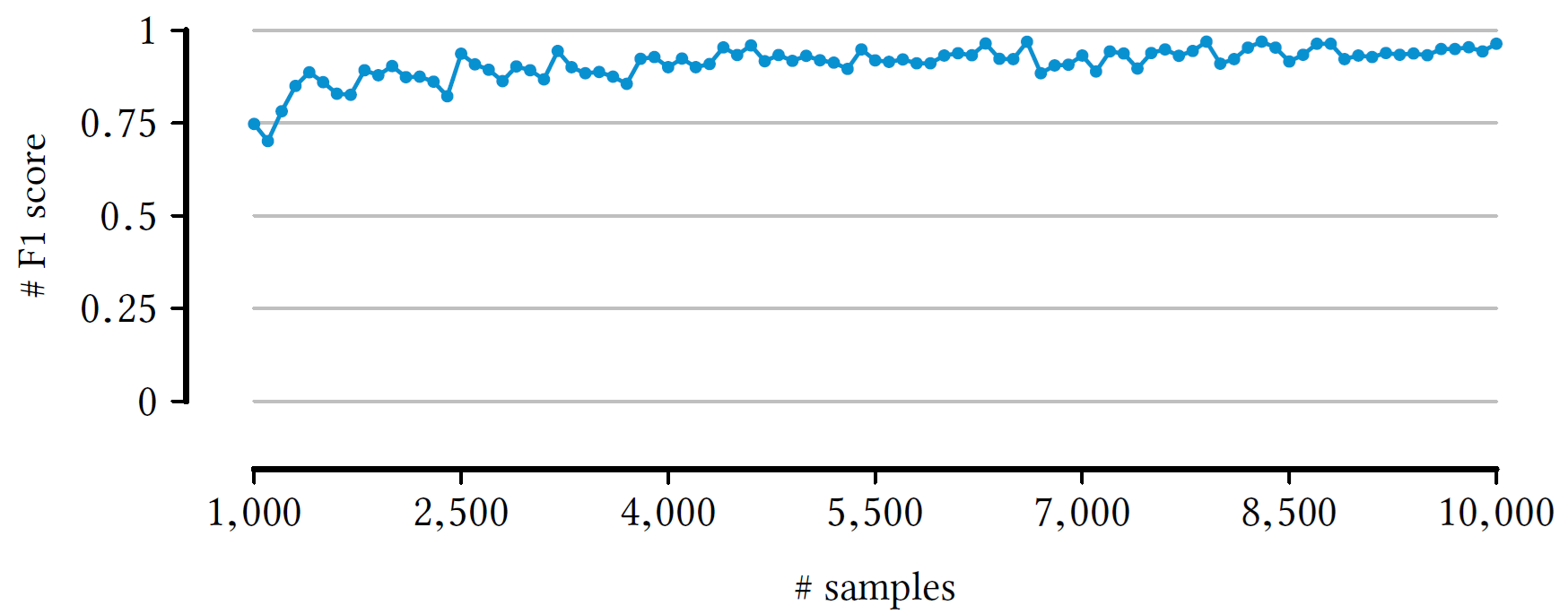
Para testar a capacidade do BinaPs de operar com poucos samples, também foi feito testes com quantidades crescentes de dados para ver sua performance.
Mesmo com um número bem reduzido de samples o BinaPs foi capaz de conseguir uma pontuação boa (Figura 5), melhorando marginalmente com mais samples até estabilizar perto do final do gráfico.
2.2.2 Reais
Foram usados 5 bases de dados reais para o comparativo entre os algoritmos:
- DNA: Dados de amplificação de DNA
- Accidents: Dados de acidentes belgas
- Instacart: Dados de compras de supermercado online
- Korsarak: Dados de cliques em um site de notícias hungaro
- Genomes: Dados de indivíduos no projeto 1000 genomes
2.2.2.1 Análise Qualitativa
Ao contrário dos dados sintéticos, não temos como saber quais padrões são “corretos” ou “incorretos”. Dessa forma, a análise é mais subjetiva. Primeiro comparamos o número de padrões encontrados (Tabela 1 (b)) nos 5 bancos de dados (Tabela 1) e o tempo de execução de cada algoritmo (Tabela 1 (c)).
| \(Dataset\) | \(\#\ rows\) | \(\#\ cols\) |
|---|---|---|
| DNA | \(2458\) | \(391\) |
| Accidents | \(340183\) | \(468\) |
| Instacart | \(2704831\) | \(1235\) |
| Kosarak | \(990002\) | \(41270\) |
| Genomes | \(2504\) | \(226623\) |
| \(Asso\) | \(BinaPs\) | \(Desc\) | \(Slim\) |
|---|---|---|---|
| \(134\) | \(131\) | \(345\) | \(281\) |
| \(133\) | \(78\) | \(215\) | \(12261\) |
| \(n/a\) | \(328\) | \(712\) | \(8119\) |
| \(n/a\) | \(302\) | \(n/a\) | \(n/a\) |
| \(n/a\) | \(42\) | \(n/a\) | \(n/a\) |
| \(Asso\) | \(BinaPs\) | \(Desc\) | \(Slim\) |
|---|---|---|---|
| \(4 m\) | \(26 s\) | \(20 s\) | \(2 s\) |
| \(12 h\) | \(6 m\) | \(14 m\) | \(21 h\) |
| \(\infty\) | \(44 m\) | \(25 m\) | \(8 h\) |
| \(\infty\) | \(5 h\) | \(\infty\) | \(\infty\) |
| \(\infty\) | \(9 m\) | \(\infty\) | \(\infty\) |
Em alguns casos, os outros algoritmos não conseguiram rodar em até 3 dias ou com 256GB de RAM. Tais cenários foram marcados com \(n/a\) ou \(\infty\) (Tabela 1 (b)).
O BinaPs retornou resultados menos redundantes, facilitando interpretabilidade. Além disso, foram notados algumas falhas em outros algoritmos, dentre eles:
- Asso não conseguiu escalar bem
- Slim encontrou milhares de resultados redundantes
- Desc sofre de underfitting e só retornou padrões de tamanho 2 no Instacart.
2.2.2.2 Análise Quantitativa
Foi feita uma análise quantitativa em 3 dos bancos de dados listados acima. Duas comparativas (DNA, Instacart) e uma individual.
DNA: BinaPs e Asso encontraram blocos de DNA e conjuntos desses blocos como estruturas, representando elementos biologicamente relevantes. Slim começa a encontrar blocos, mas faz um overfitting para padrões grandes demais que acontecem raramente e não tem estrutura evidente de blocos. Desc encontra padrões pequenos apenas graças a um underfitting.
Instacart: BinaPs encontra padrões grandes com combinações arbitrárias como um conjunto de 12 frutas comprados de formas diferentes. O Slim quebra este conjunto em milhares de padrões menores. Desc faz underfitting novamente, encontrando padrões de tamanho 2 apenas. Além disso, o BinaPs também encontrou padrões pequenos que se assemelham à lista de ingredientes de pratos culinários, mostrando relevância novamente.
Genomes: De acordo com os autores, esta seção obteve os resultados mais promissores, sendo um motivador principal para o estudo.
Foi possível encontrar padrões antes conhecidos de genes relacionados, como os NUCB2 e ABCC8 relacionados à diabetes tipo 2 e pressão alta em populações japonesas. Porém, muitas vezes esses grupos conhecidos estavam adjuntos a outros elementos, como o NCR3LG1 e ROMO1. Isso demonstra a possibilidade do uso do algoritmo para estabelecer relações novas que podem ser estudadas no futuro.
Outro exemplo foi o dos genes SF3A1, RRP7A e Z82190 onde os dois primeiros codificam proteínas que são parte do ribossomo (que por sua conta é a fábrica de proteínas da célula), já o terceiro não é caracterizado. O padrão destes juntos é uma dica que pode guiar estudos futuros nessa área.
Por final, ao analisar padrões de variantes entre alelos, o BinaPs sugere que variantes raras normalmente acompanhadas de comuns podem acontecer de um para o outro “\(0 \mid 1\)” como na literatura, mas muitas vezes também acontece em “\(1 \mid 0\)”, ou seja, os raros no alelo que antes havia comuns e vice-versa. Os autores não sabem se isso têm significado biológico, sugerindo que isso seja analisado por profissionais da área.
2.3 Execução
O artigo nos entrega um Link para o repositório. Para rodar o BinaPs em uma máquina comum é necessário:
2.3.1 Gerar dados sintéticos
- Baixar e descompactar os arquivos
- Instalar as dependências (Pytorch, Scipy, Pandas, Numpy, R)
- Alterar arquivo
genSynth.shpara parâmetros de uma máquina convencional (8 a 32GB de RAM) - Executar
./genSynth.sh
O resultado é um arquivo .dat com os dados em formato de matriz binária esparsa. Ou seja, para cada linha, os elementos da linha são os indices das posições com valor 1 na matriz original.
2.3.1.1 Executar o algoritmo
Para executar basta chamar python main.py --input <arquivo.dat> --batch_size <32 ou 64> que pode também ser adjunto dos parâmetros apresentados na Tabela 2.
| Parâmetro | Tipo | Valor padrão | Descrição |
|---|---|---|---|
--save_model |
bool |
\(False\) | Se ativado, salva o modelo treinado para disco |
--gamma |
float |
\(0.1\) | Fator de decaimento do learning rate (usado no agendador de LR) |
--lr |
float |
\(0.01\) | Taxa de aprendizado (learning rate) |
--train_set_size |
float |
\(0.9\) | Proporção dos dados usada para treinamento |
--weight_decay |
float |
\(0.0\) | Fator de penalização L2 (regularização dos pesos) |
--batch_size |
int |
\(64\) | Tamanho do batch para treinamento |
--epochs |
int |
\(10\) | Número de épocas de treinamento |
--hidden_dim |
int |
\(-1\) | Número de neurônios ocultos (usa #features se -1) |
--log_interval |
int |
\(10\) | Intervalo (em batches) para exibir logs de treino |
--seed |
int |
\(1\) | Semente para reprodutibilidade (random seed) |
--test_batch_size |
int |
\(64\) | Tamanho do batch para teste |
--thread_num |
int |
\(16\) | Número de threads a serem usadas no treinamento |
--input (-i) |
str |
Obrigatório | Caminho para o arquivo de entrada (dados usados para treino e teste) |
2.3.1.2 Saída do algoritmo
Após o treinamento marcado pelas “Epoch” temos alguns dados como a “Average loss” e a “Accuracy” seguido dos padrões dispostos na Figura 6.
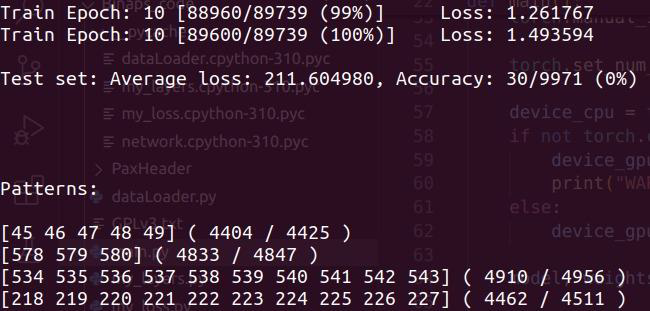
Cada linha dos padrões mostra um padrão que foi reconstruído, como por exemplo o \([45, 46, 47, 48, 49]\) seguido de dois números: O primeiro o número de amostras que ativaram todos os bits e o segundo amostras que ativaram metade dos bits do padrão. Ou seja, \(4404\) vezes os bits \(45\) a \(49\) foram completamente ativados e \(4425\) vezes parcialmente ativados (metade).
Conclusão: O padrão \([45-49]\) é altamente confiável pois aparece em mais de 99% das vezes que é parcialmente ativado, indicando forte consistência local nos dados.
3 Conclusão
O BinaPs é um algoritmo de mineração de padrões inovador, com características transparentes e eficientes em relação à competição. Seu código é aberto e bem explicado com um gerador de dados sintéticos incluso e alguns estudos preliminares de áreas que podem beneficiar do seu uso. Se for usado como uma ferramenta de forma responsável, ele pode ser um pioneiro em estudos, análises e sumarizações de dados massivos.
4 Links de Interesse
- Título: Differentiable Pattern Set Mining - Fischer e Vreeken (2021)
- Artigo: DOI, PDF, MP4 (Apresentação dos Autores)